Desmontar un receptor
Lego nucleastorm
¿Qué caracteriza a la familia de los receptores nucleares? Es decir, ¿qué tienen en común los receptores nucleares? La respuesta, en primera instancia tiene que ver con su estructura. En general, todos están codificados por genes que suelen estar formados por 8 exones, los cuales se traducen a proteínas que presentan una estructura modular. Esta organización tiene un reflejo en la organización de los intrones y exones de los genes que codifican para ellos.
En las últimas décadas se ha desentrañado la composición y el funcionamiento de estas proteínas, un logro cuyo enorme mérito puede pasarnos inadvertido.
Si nuestro cuerpo tuviera el tamaño de la Tierra, conocer cómo son y cómo funcionan los receptores nucleares equivaldría a observar y comprender cómo funciona… un smartphone o un reloj de pulsera. Si tenemos en cuenta que desde el espacio no se observa ni siquiera la muralla china, podemos hacernos una idea de hasta dónde nos ha llevado la investigación científica.

Los módulos
Los receptores nucleares comparten una estructura general básica, que puede considerarse modular: está formada por “porciones” (módulos) que desarrollan funciones diferenciadas. Las diferencias entre los diferentes miembros de la familia pueden entenderse observando la presencia o ausencia de estos módulos, así como la especificidad de cada uno de ellos.
A nivel evolutivo, la selección natural ha tenido muchas piezas sobre las que actuar (ha actuado no sólo a nivel de la proteína como un conjunto sino también a nivel de cada uno de los módulos). El refinamiento que se puede alcanzar es abrumador y no hace más que añadir interés a esta familia proteica.
Todos los receptores nucleares vienen equipados, de serie, con un módulo que les permite reconocer a moléculas concretas que les activarán (su ligandos) y, la inmensa mayoría de ellos, con un módulo que les permite reconocer secuencias concretas de DNA a las que se unirán. El módulo o dominio de unión se abrevia muchas veces como LBD (de Ligand binding domain); el dominio de unión al DNA recibe las siglas de DBD (de DNA binding domain). Estos módulos condicionan la respuesta y buena parte de la acción de los receptores nucleares.


La madeja proteínica
El funcionamiento de estos módulos y, de toda la proteína, depende de su forma; es decir, de su estructura tridimensional.
Las proteínas, químicamente, no dejan de ser una hebra de aminoácidos engarzados uno a uno; como un collar, o una cadena de maestres, forjada eslabón a eslabón, si recurrimos a un símil literario-seriéfilo.

Estas hebras se retuercen espacialmente; su plegamiento está condicionado por las atracciones y repulsiones de los elementos de la propia cadena.

Los eslabones de la cadena se van uniendo traduciendo las instrucciones que se encuentran en el RNA, el cual es una copia procesada del DNA del núcleo. Así, los eslabones de la cadena de una proteína son una traducción del código de letras de su gen (presente en el DNA).
La modularidad de los receptores nucleares no se limita sólo a su estructura tridimensional, sino que tiene su reflejo también en los genes que codifican para ellos.
En los genes de los receptores nucleares pueden distinguirse seis regiones, las cuales se han bautizado con letras mayúsculas: A, B, C, D, E y F.
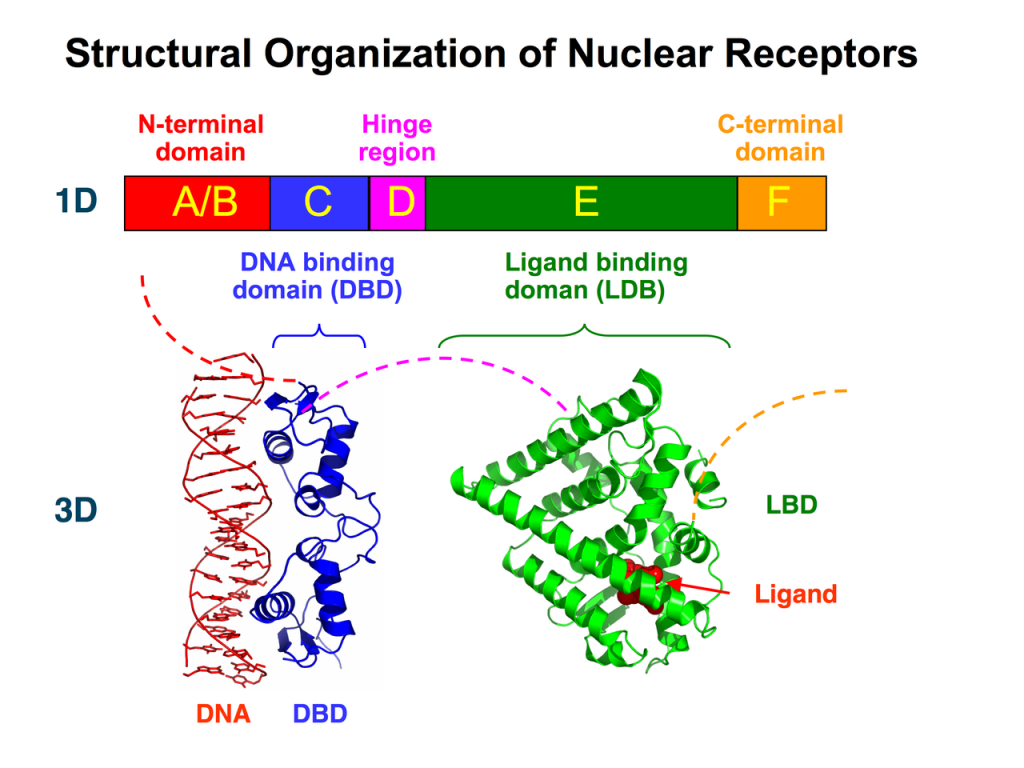
El dominio A/B se sitúa en el extremo N-terminal y es un dominio muy variable; es decir, los dominios A/B de los distintos receptores nucleares pueden ser muy distintos entre sí. Esta variabilidad ya se ve reflejada en su longitud. El dominio A/B puede contener desde 50 a 500 aminoácidos. Sea como sea, curiosamente, suele estar codificado por un solo exón. En este dominio encontramos, como mínimo, una región de transactivación, la AF-1, la cual interactúa con proteínas que determinarán la acción del receptor.
El dominio C es el dominio más conservado entre todos los receptores que lo presentan (de los 48 receptores nucleares, sólo DAX-1 y SHP carecen de él). Esta conservación implica que el dominio C, codificado en dos exones, presenta pocas diferencias entre distintos receptores nucleares. El dominio C codifica para los aminoácidos que conforman el DBD, es decir, para el módulo de reconocimiento del DNA. El DBD presenta un núcleo de 66 aminoácidos con un plegamiento característico (también altamente conservado entre receptores). Este núcleo es responsable tanto del reconocimiento de una secuencia concreta de DNA (se une sólo si encuentra un “sintagma” del DNA concreto) como de la dimerización de los receptores nucleares (muchas veces actúan en parejas). Los “sintagmas” del DNA reconocidas por los receptores nucleares reciben el nombre genérico de Hormone Response Elements (HRE) y suelen estar formados por dos “palabras” separadas por un número determinado de bases (las letras del DNA). Dentro del núcleo de 66 aminoácidos se pueden distinguir secuencias aún más cortas con funciones concretas. Así, la denominada caja P es responsable directa del reconocimiento de la palabra del HRE; la caja D, por su parte, es responsable del reconocimiento de la distancia entre las palabras del HRE.
El dominio D, poco conservado, codifica para una porción flexible de la proteína que sirve de enlace, obviamente, entre la región C y la E; entre el módulo de reconocimiento del DNA (DBD) y el de unión al ligando (LBD). Esta flexión permite a los receptores nucleares adoptar diferentes conformaciones, lo que resulta capital para que la mayoría de ellos actúen por parejas. Además, contiene una señal de localización nuclear, una especie de código postal que indica que esta proteína debe transportarse (según las condiciones) al núcleo.
El dominio E es la región más extensa. Está codificada por 5 exones y se encuentra bastante conservada. Curiosamente, su estructura secundaria (el plegamiento de los aminoácidos que la forman) en 11 o 12 hélices alfa está más conservada que la estructura primaria. Es decir, aunque puedan haber variaciones en los aminoácidos concretos que forman su secuencia lineal, estos se pliegan formando un conjunto de hélices alpha. El dominio E contiene el módulo de unión a ligando (el LBD). Es, por tanto, responsable de la especificidad fisiológica de la acción del receptor: determina en respuesta a qué estímulo actuará. Además, participa en la dimerización de los receptores, en la unión de otras proteínas que modulan su actividad (por ejemplo, coactivadores o heat-shock proteins, si vamos para nota), en su localización en el núcleo y, directamente en su activación dependiente de ligando, ya que presenta el segundo dominio de activación (AF-2).
Imágenes: Windell Oskay, Lauren Nelson, NASA Goddard, Mark Deckers, Jeff Latimer, Boghog2, Artlejandra

